Le interviste in Laboratorio
Mauro Chinappi – Blasco Morozzo della Rocca
di Pamela Pergolini
I sensori a nanoporo sono alla base di dispositivi portatili – che in genere hanno l’aspetto di una chiavetta USB – per sequenziare il DNA e hanno permesso notevoli sviluppi in genomica. Ora la prossima sfida sarà utilizzare i sensori a nanoporo per l’analisi delle proteine, molto più complesse del DNA. Ed è verso questo obiettivo che punta la ricerca a cui hanno lavorato i due docenti dell’Università di Roma Tor Vergata, Mauro Chinappi del Dipartimento di Ingegneria Industriale e Blasco Morozzo della Rocca del Dipartimento di Biologia, in collaborazione con il gruppo di ricerca guidato da Giovanni Maglia dell’Università di Groningen, in Olanda, nel Laboratorio Single-molecule biophysics.
Secondo la ricerca, pubblicata nell’ultimo numero di Nature Biotechnology
con il titolo “Translocation of linearized full-length proteins through an engineered nanopore under opposing electrophoretic force”, sarebbe possibile utilizzare i sensori a nanoporo per analizzare anche le proteine. Ad oggi, tecnologie in grado di sequenziare in modo semplice singole proteine non sono disponibili. Una tecnologia per il sequenziamento diretto di proteine potrebbe portare a una rivoluzione nella ricerca in biologia e medicina.
Per le nostre Interviste in laboratorio abbiamo chiesto agli autori della ricerca, Blasco Morozzo della Rocca, docente di Bioinformatica, e Mauro Chinappi, docente di Fluidodinamica, di raccontarci in che modo hanno lavorato insieme per cercare di capire l’efficacia di approcci ingegneristici sulla cattura e il trasporto di proteine attraverso una tecnologia innovativa come quella dei biosensori a nanoporo.
D. Qual è l’aspetto principale che lega la Biologia molecolare a questo campo dell’ingegneria, la Fluidodinamica?
R.Mauro: Nelle bionanotecnologie il confine tra le discipline è molto sfumato. In questo progetto ci occupiamo del trasporto di liquidi attraverso canali, che è tradizionalmente un argomento di cui si occupa la fluidodinamica. Nello specifico, i canali di cui ci occupiamo in questo studio sono costituiti da proteine, dunque per la loro progettazione sono indispensabili conoscenze di biologia molecolare.
R. Blasco: Nonostante il nostro inquadramento formale, abbiamo entrambi una formazione multidisciplinare e questo ci permette di poter affrontare problemi complessi con visioni complementari. È una situazione che nella scienza contemporanea si verifica sempre più spesso … fortunatamente aggiungerei.
D. Parliamo dei nanopori: quali sono le caratteristiche di questi sensori altamente innovativi, come funzionano?
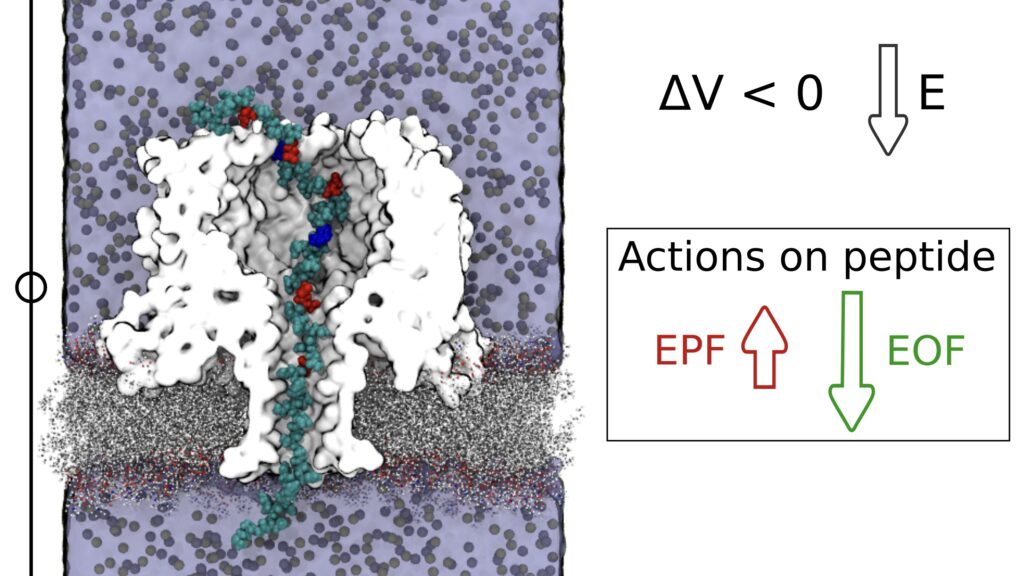
R. Mauro: Un singolo poro di pochi nanometri di diametro connette due camere in cui c’è acqua e sale. Un voltaggio applicato tra le due camere causa il passaggio di ioni da una camera all’altra. La corrente elettrica associata al passaggio di ioni può essere misurata facilmente con un amperometro. Quando una molecola è nel poro, il passaggio di ioni è ostacolato e quindi passa meno corrente elettrica nel sistema, un po’ come quando cade qualcosa in un lavandino e fluisce meno acqua attraverso lo scarico.
R. Blasco: Molecole diverse danno luogo a diversi segnali elettrici. Quindi, dalla variazione di corrente elettrica è possibile identificare la molecola che in quel momento sta occupando il poro. I sensori a nanoporo per l’analisi del DNA sono ormai una tecnologia consolidata: è molto semplice portare il DNA al poro perché è una molecola carica – e quindi è possibile guidarla con un campo elettrico – ed è anche relativamente semplice controllare il suo passaggio nel poro usando dei motori molecolari. Estendere questi approcci all’analisi di proteine è molto più complesso in quanto le proteine non hanno una carica omogenea. Una delle novità del nostro lavoro è aver mostrato come sia possibile indurre la cattura e il trasporto di proteine grazie ad un fenomeno fluidodinamico noto come elettroosmosi.
D. Che significa nella pratica “ingegnerizzare” un nanoporo biologico? Il nanoporo che avete utilizzato è stato costruito appositamente per questa ricerca?
R. Mauro: Ad oggi è possibile mutare la sequenza delle proteine per generare pori che espongano al loro interno regioni cariche positivamente o negativamente. Tuttavia, capire se e quali mutazioni sono utili per un certo obiettivo non è semplice. Le nostre simulazioni hanno permesso di comprendere in che modo le modifiche della superficie interna del poro alterino il flusso di acqua (l’elettroosmosi). Ulteriori simulazioni hanno poi permesso di quantificare le forze agenti sulla proteina all’interno del poro mostrando, ad esempio, che la forza dovuta al flusso elettroosmotico può essere così intensa da permettere di catturare e trasportare proteine anche quando la forza elettroforetica è orientata in direzione opposta.
R. Blasco: Uno dei vantaggi di usare pori biologici è che la loro struttura è determinata dagli aminoacidi che la compongono, i quali a loro volta sono codificati nel DNA che si usa per la loro produzione. In questo modo è possibile creare molte combinazioni diverse e testare il loro comportamento o efficacia rispetto a una funzione che si vuole implementare. I nostri collaboratori in Olanda, in particolare Adina Sauciuc, ne hanno prodotti oltre una decina, per cercare di capire quale andasse meglio. Incrociando i dati dei modelli, degli esperimenti e delle simulazioni abbiamo identificato le combinazioni migliori per il nostro scopo.
D. Una domanda di biologia: qual è l’utilità di poter identificare e sequenziare proteine?
R. Blasco: Le proteine sono tra gli attori principali dei fenomeni biologici, sono le operaie, le esecutrici delle più svariate funzioni, da quelle più semplici e strutturali a quelle complesse come la trasmissione di segnali nervosi o la conversione della luce in energia chimica, tanto per fare qualche esempio. Anche se spesso si dà molta importanza al DNA e ai geni (giustamente), l’informazione che essi contengono viene “messa in pratica” dalle proteine. Queste poi subiscono altre modifiche durante la loro vita, maturano con delle modificazioni chimiche, che sono spesso associate a fenomeni di regolazione e anche all’insorgenza di patologie. Potere identificare e sequenziare le proteine, con strumenti rapidi ed efficaci, avrebbe implicazioni di vasta portata anche per la diagnosi di malattie e la cura dei pazienti.
D. Questa innovativa tecnologia ha permesso di ottenere sviluppi nel sequenziamento del DNA a partire dagli anni ‘10 del 2000, quali nuove prospettive alla ricerca può aprire questo vostro studio?
R. Blasco: Ad oggi, tecnologie in grado di sequenziare direttamente singole proteine non sono disponibili. Esistono approcci che forniscono informazioni sul proteoma, ma richiedono dei passaggi complessi. In alcune tecniche le proteine vanno tagliuzzate e ricomposte, per altre servono complessi cicli di reazioni o macchinari molto sofisticati e costosi. Una tecnologia per sequenziamento diretto di proteine potrebbe portare a un cambio di passo nella ricerca in biologia e medicina forse paragonabile a quel che è accaduto qualche decade fa con la disponibilità di sequenziatori di DNA a basso costo, i cui riflessi e ricadute si stanno raccogliendo anche ora.
R. Mauro: Il nostro studio è un tassello che potrebbe aiutare a risolvere uno dei problemi principali dei sensori a nanoporo per le proteine: la possibilità di controllare il trasporto delle proteine attraverso il poro. Fino a qualche anno fa, solo pochi gruppi di ricerca studiavano la possibilità di usare approcci nanofluidici come l’elettroosmosi per controllare il trasporto di proteine. Ora vari gruppi si stanno muovendo in questa direzione e, il nostro studio, in qualche forma, suggerisce che questa sia una direzione promettente.
D. In quali altri campi possono essere utilizzati i sensori a nanoporo?
Blasco: Ovunque siano coinvolti attori biologici! Oltre alla medicina, la microbiologia e l’ambiente mi vengono in mente tutti quei processi industriali che coinvolgono organismi, come la produzione di yogurt, vino e birra.
 LE PAROLE DELLA SCIENZA
LE PAROLE DELLA SCIENZA
Le parole del giorno
ELETTROOSMOSI: trasporto di acqua indotto da un campo elettrico esterno, da qui il nome elettroosmosi, dal greco ὠσμός “spinta, impulso”. Immaginiamo, ad esempio, un canale sulle cui pareti ci siano, cariche fisse negative e supponiamo che in questo canale ci sia una soluzione elettrolitica (acqua e sale). Queste cariche fisse sulle pareti del canale attireranno gli ioni positivi presenti in soluzione. A questo punto avremo all’interno del canale una prevalenza di ioni positivi. Sotto l’azione di un campo elettrico esterno, questi inizieranno a muoversi e trascineranno l’acqua.
ELETTROFORESI: movimento di una particella o molecola carica indotto da un campo elettrico esterno. È un fenomeno che si usa molto, ad esempio, nelle analisi biochimiche per muovere e separare molecole (proteine, DNA) per poi identificarle.